허혈성 뇌졸중의 진단, 치료 및 예후 예측에 대한 기계 학습의 응용: 서술적 고찰
Machine learning application in ischemic stroke diagnosis, management, and outcome prediction: a narrative review
Article information
Trans Abstract
Stroke is a leading cause of disability and death. The condition requires prompt diagnosis and treatment. The quality of care provided to patients with stroke can vary depending on the availability of medical resources, which in turn, can affect prognosis. Recently, there has been growing interest in using machine learning (ML) to support stroke diagnosis and treatment decisions based on large medical data sets. Current ML applications in stroke care can be divided into two categories: analysis of neuroimaging data and clinical informationbased predictive models. Using ML to analyze neuroimaging data can increase the efficiency and accuracy of diagnoses. Commercial software that uses ML algorithms is already being used in the medical field. Additionally, the accuracy of predictive ML models is improving with the integration of radiomics and clinical data. is expected to be important for improving the quality of care for patients with stroke.
서 론
뇌졸중은 뇌혈관 문제로 인한 뇌 손상에 의해 신경학적 증상이 발생하는 것으로 정의되며[1], 전 세계적으로 사망과 장애를 유발하는 주요 원인 중 하나이다[2]. 고령화가 진행되면서 국내 뇌졸중 유병률은 점차 증가할 것으로 예상된다. 건강보험심사평가원의 국민관심질병통계에 따르면 2021년에만 약 62만 명의 환자가 뇌졸중으로 진료를 받았고, 이에 따른 요양급여비용은 약 2조 4천억 원이 소요되었으며, 이러한 비용은 계속해서 증가하고 있다. 따라서 뇌졸중을 효과적으로 진단하고 치료하여 환자의 예후를 개선시키는 것은 개인과 사회경제적 부담을 줄이는 데 매우 중요하다.
뇌졸중이 발생하면 단시간 내에 뇌손상이 진행되며, 한번 손상된 뇌는 완전히 회복되기 어렵기 때문에[3] 뇌졸중을 빠르고 정확하게 진단하고 적절한 급성기 치료를 시행하는 것이 필요하다. 뇌 영상 검사는 뇌졸중의 진단과 분류에 필수적이며, 환자의 예후를 예측하는 데에도 중요한 역할을 한다[4,5]. 하지만 뇌 영상 검사 기법의 발전과 다양화로 인해 영상 촬영, 후처리 및 판독에 더 많은 시간이 소요될 수 있어 신경 영상 검사의 시행 및 해석에 걸리는 시간은 환자의 치료를 지연시키는 요인 중 하나가 될 수 있다[6]. 또한 신경 영상 검사는 단순 방사선 촬영과 달리 주로 computed tomography (CT)나 magnetic resonance imaging (MRI)을 사용해서 촬영하는 경우가 많고, 검사 부위의 해부학적 구조가 복잡하기 때문에 의사의 판독 숙련도가 검사 결과의 정확도에 영향을 미칠 수 있으며, 판독자 간의 해석 차이가 발생할 수 있다[7,8]. 뇌졸중 진단과 치료는 뇌 영상 검사 결과 외에도 환자의 임상 양상 및 동반 질환과 같은 다양한 인자들을 고려해야 한다. 따라서 의료 자원이 부족한 지역에서는 전문 의료 인력 및 검사 장비의 부족으로 인해 뇌졸중 환자 진료의 질에 부정적인 영향을 미칠 수 있으며, 결과적으로 환자의 예후에 악영향을 끼칠 수 있다.
최근에는 기계 학습이 의료 서비스 분야에서 광범위하고 방대한 의료 데이터를 기반으로 다양한 질병의 진단, 예후 예측 및 치료 계획 결정에 도움을 주고 있다. 복잡한 의료 데이터의 특성을 이해하고 분석할 때 전통적인 통계적 방법론이 제한적일 수 있는 반면, 기계 학습은 매우 효과적인 도구로 활용될 수 있다. 이러한 기술은 고차원적이고 비선형적인 패턴을 탐지하고, 질병의 복잡한 원인과 결과 간의 관계를 이해하는 데 도움을 주며, 임상 현장에서는 의료 데이터를 기반으로 개인화된 치료를 제공하는 데 효과적으로 활용될 수 있다. 기계 학습을 통한 질환의 진단 보조(computer-aided diagnosis)는 뇌졸중과 같이 빠른 시간 내에 치료가 필요하고 발생률이 높으며 진료의 질에 따라서 환자의 예후가 크게 달라질 수 있는 응급 질환에서 특히 중요하다. 따라서 뇌졸중 진료 분야에서는 기계 학습을 활용한 다양한 연구가 진행되고 있으며, 실제 임상에서의 적용도 점점 더 증가하고 있다.
뇌졸중 분야에서 기계 학습은 다양한 영역에서 연구되고 있다. 대표적으로 신경 영상 분석을 통해 뇌졸중 병변을 검출(detection) 및 분할(segmentation)하고[9-11] 발생 기전에 따라 분류(classification)하며[12] 큰 대뇌혈관의 폐색을 감지하여 재관류 치료 대상자를 선별하고[13,14] 치료 합병증을 예측(prognostication) [15,16]하는 연구 등이 진행되었다. 또한 다양한 임상 변수들을 이용하여 뇌경색 환자의 치료 반응과 임상적 예후를 예측하고자 하는 연구도 이루어지고 있으며, 최근에는 영상 결과와 임상 변수를 함께 분석하여 보다 정확한 예측을 시도하는 연구가 진행되고 있다[17].
본 종설에서는 주로 급성 뇌경색 환자의 진료 과정에서 기계 학습의 적용에 관한 최근 연구와 그 근거에 대해 고찰하고, 기계 학습을 활용한 뇌졸중 진료의 발전 방향 및 한계에 대하여 논의하고자 한다.
본 론
1. 기계 학습의 개요
기계 학습 모델의 개발은 지도 학습(supervised learning) 또는 비지도 학습(unsupervised learning)을 통해 이루어질 수 있다[18]. 지도 학습은 전문가가 학습 데이터를 레이블링(labeling)하고, 레이블링된 데이터를 바탕으로 정해진 알고리즘에 의해 기계 학습 모델을 학습시키는 방식이다. 의료 분야에서 데이터 레이블링은 주로 영상 분류 또는 병변 부위(region of interest)를 수동으로 분할하는 작업으로 이루어진다. 이 작업은 상당한 시간과 노력이 필요하고, 데이터 레이블링의 정확도가 모델의 성능에 큰 영향을 미칠 수 있다. 그러나 지도 학습을 통해 개발된 모델은 사람이 식별한 정답을 기반으로 학습하므로 모델의 결과를 이해하고 해석하기 쉽다는 장점이 있다. 지도 학습 알고리즘의 대표적인 예로는 linear regression, logistic regression, support vector machine, decision trees, random forest, naïve Bayes, K-nearest neighbor, artificial neural networks 등이 있다[19]. 특히 딥러닝(deep learning)은 artificial neural network에 기반한 기계 학습의 모델링 방법 중 하나로, 여러 개의 은닉층을 사용함으로써 간단한 패턴에서 복잡한 패턴까지 계층적으로 학습이 가능하다[20]. 딥러닝은 대량의 데이터를 기반으로 복잡한 문제를 해결하는 데 강점이 있지만 전통적인 기계 학습에 비해 데이터 의존도가 높기 때문에 불균형한 분포를 보이는 데이터로 학습하게 되면 모델의 성능에 영향을 줄 수 있는 long-tail problem이 발생할 수 있고[21], shortcut learning으로 인해 학습된 상황 이외의 실제 사례에서 성능이 저하되는 제한점도 있다[22]. 그러나 활용 가능한 빅데이터가 많아지고 컴퓨팅 파워가 발전하면서 의료 영상, 자연어 처리 등 다양한 분야에서 그 중요성이 커지고 있다.
반면에 비지도 학습을 기반으로 한 영상 분석 모델은 데이터 레이블링 과정 없이 데이터의 핵심 특징을 추출하고 패턴을 학습하여 이상을 감지하거나 패턴에 따라 분류하는 데 활용된다. 의료 영상 분야에서는 사람의 시각으로는 식별하기 어려운 질감(texture), 균질성(homogeneity)과 같은 특징들을 자동으로 감지하여 분석하는 데 큰 장점을 가지며, 지도 학습에서 필요한 적절한 특징을 찾기 위한 전처리 과정으로 활용될 수 있다. 비지도 학습에 의한 알고리즘으로는 K-means clustering, probabilistic clustering, hidden Markov model, hierarchial clustering, linear discrimination analysis 등이 있다[19].
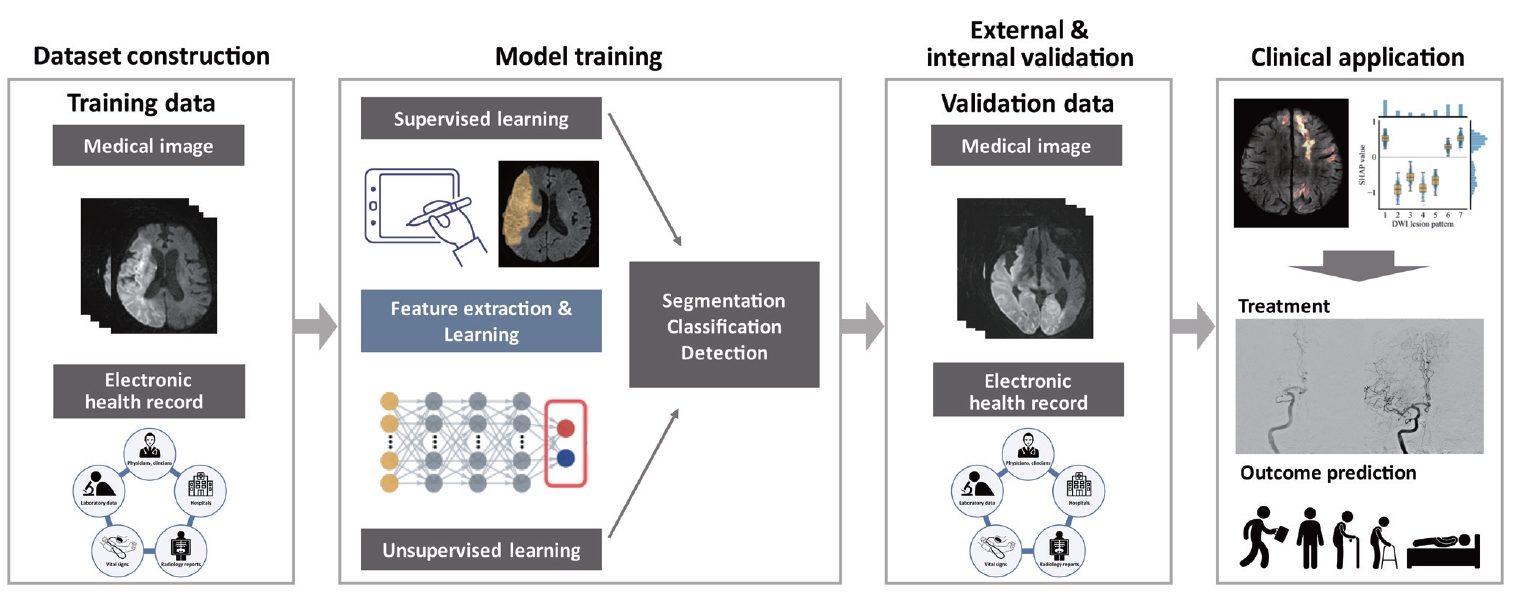
다양한 학습 방법을 통해 개발된 모델은 학습에 사용되지 않은 별도의 데이터셋에서 검증(validation)을 통해 모델의 성능을 확인하는 과정을 거친다. 이러한 과정은 과적합(overfitting)을 방지하고[23], 모델이 학습 데이터뿐만 아니라 다른 독립된 데이터에서도 효과적으로 작동하는지 확인하는 데 중요하다. 모델이 충분히 견고하고 정확하게 예측할 수 있다는 것을 검증하면 새로운 데이터에 적용하여 예측을 수행할 수 있으며, 결과적으로 개발된 기계 학습 모델이 의료 분야에서 실제로 적용되어 질병 진단, 예후 예측 등의 중요한 의사결정을 지원하는 데 기여하게 된다(Fig. 1). 최근에는 의료 현장에서 기계 학습을 이용한 예측 모델의 안전하고 효율적인 사용을 위해 데이터 준비, 모델 개발과 검증, 소프트웨어 개발, 모델의 영향 평가 및 실제 적용에 관한 다양한 지침과 질 관리 표준들이 출판되고 있다[24].
2. 뇌경색 환자에서 기계 학습을 이용한 신경 영상 분석
의료 영상 분야에서 기계 학습은 주로 촬영 시간 단축, 영상 최적화 및 품질 향상, 영상 분석을 통한 환자 분류 및 작업 흐름 개선, 진단 보조 및 치료 방향 결정에 활용될 수 있다. 뇌졸중 환자의 경우 영상 검사 시 환자의 협조가 어려울 때가 많아 촬영 시간 단축 및 운동 인공물(motion artifact)과 잡음(noise) 등을 최소화하여 최적화된 영상 품질을 유지 및 확보하는 것도 기계 학습의 중요한 용도 중 하나이다[25]. 그러나 임상의의 관점에서 뇌졸중 환자의 진료에 기계 학습을 활용하는 주된 목적은 자동화된 영상 분석을 통해 뇌졸중 진단의 속도와 정확도를 향상시키고, 이를 통해 진단부터 치료까지의 작업 흐름을 개선하여 환자의 예후를 향상시키는 것이다. 기존의 뇌졸중 환자 진료에서는 신경 영상 결과를 영상의학전문의 또는 임상의가 주로 주관적인 방법으로 해석하고, 그에 따라 치료가 이루어져 왔다. 그러나 최근에는 기계 학습 발전으로 이러한 과정을 자동화하고 정량적으로 분석하는 것이 가능해지고 있다.
신경 영상 분석을 위해서는 영상의 전처리 과정이 필요하다. 이는 영상 결과에서 분석에 사용할 영역을 감지하고(field of view detection), 두개골을 제거하여(skull stripping) 공통공간에 등록(registration)하는 과정으로 이루어져 있다. 영상의 전처리는 FMRIB Software Library [26], FreeSurfer [27] 등 다양한 소프트웨어를 사용하여 수행할 수 있지만, 이는 상당한 시간이 소요되는 작업이기 때문에 최근에는 기계 학습을 통해 전처리를 시행하거나, 영상 등록 과정을 생략하고 분석을 진행하는 방법이 도입되고 있다.
본 섹션에서는 주로 신경 영상을 활용하여 뇌졸중의 진단 및 분류, 병변 분할 및 최종 병변 예측, 합병증과 임상적 예후 예측에 기계 학습이 어떻게 활용될 수 있는지에 대하여 설명하고자 한다.
1) 뇌경색 진단 및 분류(stroke diagnosis and classification)
뇌졸중 환자의 진단은 뇌졸중과 뇌졸중 유사 질환(stroke mimics)의 구분에서 시작된다. 뇌졸중이 의심되어 응급실로 내원한 환자 중에서 40% 이상의 환자와[28] 혈전용해 치료를 받은 환자 중 13.8%의 환자가 뇌졸중 유사 환자로 보고되었다[29]. 따라서 뇌졸중이 의심될 때, 가장 빠르고 쉽게 시행할 수 있는 뇌 CT 영상을 사용하여 이상 여부를 스크리닝하고자 하는 연구들이 진행되었다[30,31]. Titano 등[31]은 3D 컨볼루션 신경망(3-dimensional convolutional neural network, 3D-CNN) 아키텍처를 사용하여 37,000여 명의 뇌 CT 영상과 의사 판독 결과, 전자 의무 기록을 학습하여 뇌 CT에서 뇌출혈, 뇌경색, 수두증과 같은 중대한 이상 여부를 분류하는 모델을 개발했다. 이 모델을 사용한 임상시험 결과, 긴급한 케이스에 대해 사람이 진행한 판독 및 환자 분류보다 150배 빠르게 알림을 보낼 수 있었다.
뇌 CT 영상에서 초기 허혈성 변화를 확인하고 뇌경색을 진단하는 것은 쉽지 않다. 따라서 비조영증강 CT에서 뇌경색 여부를 확인하고자 하는 여러 연구들이 진행되었다[32-34]. Sahoo 등[32]은 증상 발생 후 12시간 이내의 뇌경색이 있는 환자와 정상 환자의 비조영증강 CT 영상을 사용하여 급성기 뇌경색 여부를 구별하는 딥러닝 모델을 만들었고, 해당 모델은 Visual Geometry Group 16의 아키텍쳐를 사용하여 area under the receiver operating characteristic curve (AUC) 0.83, sensitivity 0.85, specificity 0.82로 급성 뇌경색 여부를 식별할 수 있었다. Lu 등[33]은 급성 뇌경색이 의심되지만 비조영증강 CT에서 병변이 확인되지 않는 환자에서 병변의 위치를 특정하고 분류하는 2단계 딥러닝 모델을 만들었다. 모델의 성능은 AUC 0.84로 영상의학전문의보다 정확도가 높았고, 외부 검증 코호트에서 영상의학전문의의 진단 정확도를 향상시켰다. 전방 순환계 뇌경색과 비교하여 후방 순환계 뇌경색은 CT 상에서 허혈성 병변을 확인하는 것이 상대적으로 어렵지만, Kniep 등[34]은 random forest 알고리즘을 이용하여 소뇌(AUC, 0.7) 및 시상 병변(AUC, 0.82)을 초기에 탐지할 수 있다고 보고하였다. 그러나 해당 모델의 성능은 전방 순환계 뇌경색 연구에 비해 상대적으로 낮았다.
뇌경색 환자에서 영상 정보는 뇌경색의 병인을 분류하거나 추정하는 데에도 도움을 줄 수 있다. MRI-GENIE 데이터를 기반으로 한 연구에서는 3D-CNN을 활용하여 확산강조 MRI에서 뇌경색의 위치와 부피를 분석하였다. 그 결과 소혈관폐색으로 인한 뇌경색은 특정 호발 위치에서 주로 발생하며, 뇌경색의 부피가 작아 다른 원인의 뇌경색과 차이를 보였다[35]. Jeong 등[36]은 뇌경색 환자의 흉부 엑스레이를 활용하여 심장색전성 뇌경색을 상대적으로 높은 성능(AUC, 0.86)으로 분류하는 등, 뇌졸중을 진단하고 분류하는 데 있어 기계 학습을 활용한 다양한 시도가 진행되고 있다.
2) 재관류 치료 대상자 식별(identification of reperfusion therapy candidate)
재관류 치료는 뇌경색 환자의 증상을 개선하고 예후를 호전시킬 수 있는 중요한 치료 방법으로, 뇌경색 증상이 발생한 후 4.5시간 이내에 정맥혈전용해제 치료를 받는 환자에게 효과가 있음이 잘 알려져 있다. 그러나 전체 뇌경색 환자 4명 중 1명은 증상 발생 시각이 명확하지 않아 혈전용해 치료를 받지 못한다[37]. 따라서 기계 학습을 통해 MRI 영상을 분석하여 증상 발생 시각이 혈전용해 치료가 가능한 4.5시간 이내에 해당하는지 구분하는 연구들이 진행되었다[38-41]. Lee 등[38]이 수행한 연구에서 기계 학습을 이용한 알고리즘은 사람의 판단보다 더 높은 sensitivity (75.8 vs. 48.5)와 유사한 specificity (82.6 vs. 91.3)를 나타내어 정맥혈전용해제 사용 대상자를 선별할 때 기계 학습의 적용 가능성을 확인할 수 있었다.
뇌경색 중 약 1/3이 큰 혈관 폐색에 의해 발생하며[42,43], 급성 뇌경색 환자에서 기계 학습을 이용하여 큰 혈관 폐색을 감지하는 것은 기계적 혈전제거술이 필요한 환자를 선별하고 치료가 가능한 병원으로 전원하는 데 실질적인 도움을 줄 수 있다. Stib 등[44]은 DenseNet-121 아키텍처를 적용하여 혈관조영 CT에서 큰혈관폐색을 확인하였으며, 다중 시기 혈관조영 CT가 단일 시기 혈관조영 CT에 비해 우수한 결과를 보였다(AUC, 0.89 vs. 0.74). Czap 등[45]은 DeepSymNet-v2 알고리즘을 이용하여 mobile stroke unit에서 촬영한 혈관조영 CT에서도 큰혈관폐색을 AUC 0.80의 정확도로 감지하여 병원 전단계에서의 적용 가능성을 입증하였다.
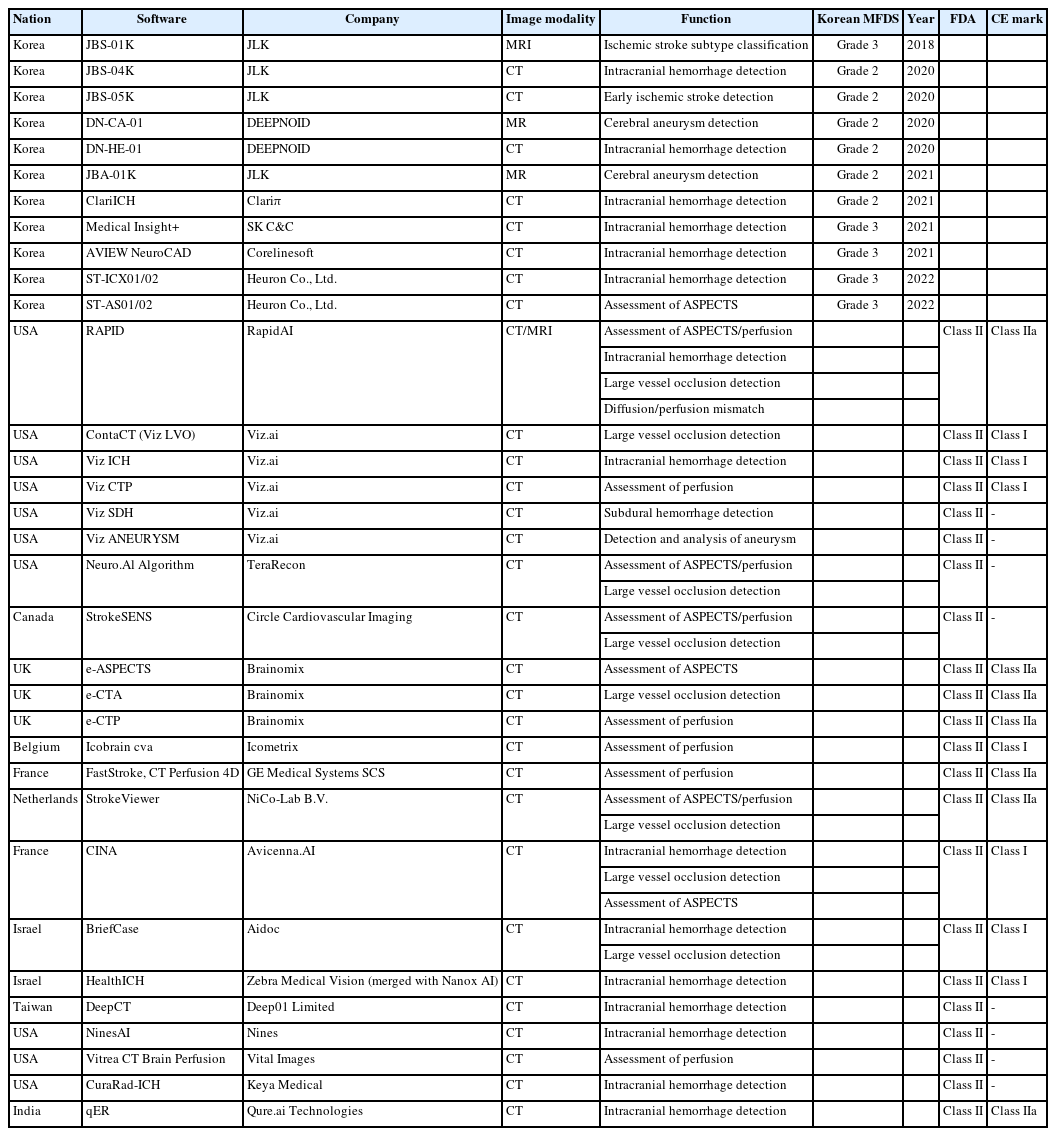
증상 발생 후 재관류까지 걸리는 시간은 환자의 예후에 큰 영향을 미치기 때문에[46] 고전적인 알고리즘 혹은 기계 학습을 이용하여 혈관조영 사진을 자동으로 분석하고 작업 흐름을 개선시키기 위해 Viz LVO (Viz.ai, San Francisco, CA, USA), Rapid LVO (RapidAI, Menlo Park, CA, USA), CINA (Avicenna.ai, La Ciotat, France), BriefCase (Aidoc, Tel Aviv, Israel), StrokeViewer (NiCo-Lab B.V., Amsterdam, Netherlands), StrokeSENS LVO (Circle Cardiovascular Imaging, Calgary, Canada), e-CTA (Brainomix, Oxford, UK) 등 다수의 소프트웨어가 상용화되어 있다(Table 1). 큰혈관폐색 진단의 경우 높은 민감도가 중요하며, 상용화된 소프트웨어는 큰혈관폐색을 감지하는 데 높은 민감도와 정확도를 보이는 것이 여러 연구에서 확인되었다[13,47-52]. 이러한 소프트웨어는 진료 및 치료 시간을 단축시키는 임상 효용을 입증하였으며, 실제 진료 현장에서도 활발하게 사용되고 있다[14,53,54].
최근에는 뇌 영상을 분석하여 기계적 혈전제거술이 도움이 될 만한 환자를 선별하고자 하는 연구들도 진행되었다. Gao 등[55]은 뇌관류 CT 및 MRI 영상의 매개변수를 기반으로 학습시킨 CNN 분류 모델을 사용하여 기계적 혈전제거술이 가능한 시간인 6시간 이내의 환자를 AUC 0.935의 상대적으로 높은 정확도로 선별할 수 있음을 보고하였다. 또한 Kim 등[56]은 병변의 부피, 크기, 겉보기확산계수(apparent diffusion coefficient, ADC) 비율 분포 등 확산강조 MRI의 영상 특성만을 이용하여 확산-관류 불일치에 의한 예측보다 높은 정확도로 좋은 임상적 예후(90일 modified Rankin Scale [mRS], 0-2)를 예측할 수 있음을 보여주었다.
RAPID 소프트웨어를 사용한 DEFUSE 3와 DAWN 연구 이후로 허혈중심부와 허혈반음영의 차이를 확인하여 증상 발생 후 24시간까지 기계적 혈전제거술을 시행할 수 있게 되면서, 이에 대한 평가와 적절한 환자 선정의 중요성이 강조되고 있다[57,58]. 이에 확산-관류 불일치를 평가하는 다양한 소프트웨어들이 개발되어 임상 현장에서 사용되고 있으나 소프트웨어 종류에 따라 결과가 다를 수 있으며, 이는 환자의 기계적 혈전제거술 시행 여부에 영향을 미칠 수 있다[59,60]. 최근에는 딥러닝을 사용하여 확산강조 MRI와 뇌관류 MRI 상의 불일치를 평가하고 예측하는 다양한 연구들이 진행되었고, 이는 기존의 알고리즘 및 임상 증상-확산강조 MRI 불일치 결과보다 우수한 성능을 나타내고 있다[61,62].
측부순환(collateral circulation)은 기계적 혈전제거술 시행 환자의 예후에 중요한 영향을 미치며, 시술 여부를 결정하는 데 중요한 고려 인자이다[63]. 고전적인 측부순환 평가는 주로 시각적으로 등급을 나누는 방법을 사용하는데, 판독자 간의 차이가 날 수 있고 정량화가 어려운 한계가 있다. 정성적인 측부순환 평가를 위해 상용화된 소프트웨어로는 StrokeViewer와 e-CTA가 있으며, 이러한 소프트웨어를 사용한 측부순환 평가는 시각적 평가와 비교하여 중간 정도의 일치도를 보였고 소프트웨어의 지원을 받는 경우 판독 시간이 감소하고 판독자 간의 일치도가 증가하였다[64,65]. 또한 측부순환의 정량적 평가를 위해 Boers 등[66]이 진행한 연구에서 혈관조영 CT에서 허혈성 병변 위치의 혈관을 분할하고 반대편 영역과 비교하여 측부순환을 정량화한 결과는 시각적 평가 결과와 좋은 상관관계를 나타내며(r=0.75), 기계적 혈전제거술의 효과에 영향을 미치는 것을 확인하였다.
3) CT 및 MRI를 이용한 뇌경색 병변 분할(lesion segmentation)
급성기에 시행한 비조영증강 CT에서 초기 허혈성 변화는 변화 정도가 작고 경계가 명확하지 않아 병변 분할이 쉽지 않다. Alberta stroke program early CT score (ASPECTS)는 비조영증강 CT에서 전방 순환계에 발생한 초기 허혈성 변화의 정도를 확인할 수 있는 반정량화된 점수 체계로 널리 사용되고 있지만, 판독자 간의 일치도가 높지 않고 판독자의 숙련도에 따라 결과가 다르게 나타날 수 있는 한계가 있다[67,68]. 이러한 한계를 극복하기 위해 Kuang 등[69]은 뇌경색 환자의 비조영증강 CT에서 각 ASPECTS 영역에 대한 영상 특성을 추출하고 분류하여 random forest 방법으로 학습한 모델을 개발하였고, 영상 전문가가 평가한 확산강조 MRI 영상의 ASPECTS와 비교하여 클래스 내 상관계수 0.76으로 비교적 높은 일치도를 보였다. ASPECTS의 정확한 평가는 기계적 혈전제거술 시행 여부를 결정하는 데 중요하기 때문에 Rapid ASPECTS (RapidAI), Heuron ASPECTS (Heuron Co., Ltd., Incheon, Korea), e-ASPECTS (Brainomix), syngo.CT ASPECTS (Siemens Healthineers, Erlangen, Germany), CINA-ASPECTS (Avicenna.AI) 등과 같은 자동화된 ASPECTS 평가 소프트웨어가 개발되었다. 이러한 자동화된 소프트웨어를 사용하여 평가한 ASPECTS는 영상의학전문의가 평가한 ASPECTS와 초기 및 추적 CT 영상에서 높은 일치율을 보였고[70], 판독자의 ASPECTS 평가 능력을 향상시키는 결과를 보였다[71]. 그러나 영상 전문가 및 소프트웨어가 평가한 ASPECTS 사이의 일치율은 대략 0.7-0.8 정도로 사용된 프로그램과 연구에 따라서 차이가 있다[72-74].
비조영증강 CT를 활용하여 뇌경색 병변을 직접 분할하는 연구로 Chen 등[75]은 mirror assembly module을 디자인하고, 포괄적 경로(global path)와 국소 경로(local path)를 갖는 새로운 deep CNN을 개발하여 병변을 분할하였으며, 이를 통해 ASPECTS를 자동으로 평가하였다. CNN을 이용한 병변 분할은 영상의학전문의가 수동으로 시행한 병변 분할 결과와 비교하여 높은 일치도를 보였다(dice score coefficient [DSC], 0.762; AUC, 0.729).
또한 비조영증강 CT에서 U-Net 아키텍쳐를 활용하여 확산강조 MRI에서 확인되는 초기 허혈성 변화를 감지하고, 허혈성 병변의 부피를 정량적으로 평가하는 연구들도 진행되었다(Table 2). 이러한 연구에서는 딥러닝 모델을 활용하여 급성 뇌경색의 존재 여부를 확인하고, 치료에 영향을 미칠 수 있는 크기가 큰 뇌경색을 구분하며, 병변 위치를 정확하게 확인할 수 있었다[76]. 또한 뇌경색의 부피를 측정하는데 있어 확산강조 MRI 영상과 높은 상관관계를 보였다(r=0.76) [77].
MRI는 CT에 비해 급성 뇌경색 병변을 시각적으로 구별하기 쉬운 장점이 있으며, 특히 확산강조 MRI는 허혈중심부를 평가하는 표준 영상으로 간주되고 있다. 확산강조 MRI의 b-value 1000 (b1000) 영상은 뇌경색을 감지하는 민감도가 높지만, 시간에 따라 신호강도가 변화할 수 있고 T2 shine-through 인공물 효과가 발생할 수 있어서 초기 상업용 알고리즘은 주로 ADC 값 620×10-6 mm2/s를 기준으로 허혈중심부를 정의하였다[78]. 그러나 최근 연구에서는 CNN 기법을 포함한 딥러닝 알고리즘을 사용하여 b1000 영상과 ADC 영상에서 허혈중심부를 분할하고 부피를 평가하는 데 있어서 상업용 알고리즘과 비슷하거나 더 높은 정확도를 보여주고 있다[10,79-81].
Winzeck 등[10]은 확산강조 MRI (b1000, b0) 및 ADC 영상을 이용하여 CNN 앙상블 모델을 개발하였으며, 개별 CNN 모델보다 높은 정확도를 달성하였다. 이 모델을 활용하여 측정한 뇌경색 부피는 수동으로 분할한 결과와 높은 상관관계를 보였다(r=0.91). 또한 확산강조 MRI 영상에서 U-Net과 DenseNet을 활용하여 뇌경색 병변을 분할한 연구에서 CNN 알고리즘은 ADC 신호강도를 기준으로 한 전통적인 알고리즘보다 수동으로 분할한 병변과 높은 일치도를 보였다(DSC, 0.86) [80].
4) 뇌경색 최종 병변 예측(prediction of final infarct lesions)
뇌경색 환자에서 허혈성 병변의 크기와 위치는 환자의 예후에 직접적으로 영향을 미치는 가장 중요한 인자 중 하나이다. 일반적으로 뇌경색 병변에서 허혈중심부는 비가역적인 손상이 진행되어 재관류 치료를 하더라도 회복이 되지 않을 병변을 의미한다[82]. 따라서 증상 발생 초기에 허혈중심부를 예측하는 것은 재관류 치료 여부를 결정하고, 환자의 예후를 예측하는 데 중요하다.
뇌관류 CT는 다양한 매개변수에 대한 정량화된 정보를 제공한다. 이 정보를 이용하여 허혈중심부 및 허혈반음영 영역을 정의할 수 있지만, 확산강조 MRI처럼 육안으로 허혈중심부를 구분하는 것은 어렵다. 따라서 기계 학습을 활용하여 허혈중심부와 반음영을 판별하는 Rapid CTP (RapidAI), Viz CTP (Viz.ai), F-STROKE (Neuroblem Technology, Shanghai, China), MIStar (Apollo Medical Imaging Technology, Melbourne, Australia), Olea Sphere (Olea Medical, La Ciotat, France) 등의 다양한 소프트웨어가 개발되었으며, 이 중에서 널리 사용 중인 프로그램이 Rapid CTP이다[83-85]. Rapid CTP는 relative cerebral blood flow (rCBF) 30% 미만을 허혈중심부, time-tomaximum (Tmax) 6초 초과를 허혈반음영으로 정의하며, 다양한 임상시험에서 허혈중심부 및 허혈반음영을 정의하는 데 사용되었다[57,58,86,87]. 그러나 허혈중심부는 증상 발생 후 뇌관류 영상을 시행하기까지 소요 시간 등에 따라 달라질 수 있으며[82], CBF 등 단일 매개변수의 임계값에 기초한 허혈중심부 및 반음영의 예측은 정확도 면에서 한계가 있다[81].
따라서 최근에는 딥러닝을 활용하여 뇌관류 CT의 다양한 매개변수 또는 deconvolution하지 않은 원본 데이터를 시간 변수와 함께 이용하여 허혈중심부 및 반음영 영역을 정의하는 연구들이 진행되었다[81,88-92]. 예를 들어, 2018년에 진행된 ischemic stroke lesion segmentation (ISLES) 챌린지에서는 뇌관류 CT 데이터를 활용하여 확산강조 MRI에서 관찰되는 허혈중심부를 추정하는 과제가 있었으며, 다양한 기계 학습 모델이 사용되었다. 이 중에서 성능이 가장 좋은 모델은 DSC가 0.51이며, 절대 부피 차이는 10.1 mL로 보고되었다[88]. 이러한 연구들은 기존의 임계값 기준 방식보다 우월한 결과를 제시하였으며, 관련 분야에서 연구가 활발하게 진행되고 있다.
초기 확산강조 MRI에서 관찰되는 뇌경색 병변은 시간이 지나면서 일부 회복되거나 반대로 진행할 수도 있다. 급성기 확산강조영상, ADC 영상, 임계 ADC 지도를 사용하여 3-7일째 추적 관찰된 대뇌 영상을 기반으로 최종 뇌경색 병변을 예측한 연구에서 3D attention gated (AG) U-Net을 사용한 CNN 모델이 매우 높은 정확도(AUC, 0.91)를 보였다[93]. 그러나 재관류 치료를 받은 환자에서는 치료받은 혈관의 재관류 정도가 최종적인 뇌경색 병변 크기에 영향을 미칠 수 있고, 이에 따라 모델의 예측 성능에 제한이 있을 수 있으므로 이에 대한 연구가 필요하다. Yu 등[94]은 imaging collaterals in acute stroke (iCAS) 연구 및 DEFUSE-2 연구에서 획득한 급성기 확산강조 MRI에서 3D AG U-Net 아키텍처를 사용하여 최종 병변을 추정하는 딥러닝 모델을 개발하였다. 이 모델은 3-7일째 추적 관찰한 fluid-attenuated inversion recovery MRI 영상의 최종 병변을 AUC 0.92, DSC 0.53으로 예측할 수 있었다[94]. 또한 재관류 성공 여부에 따라 시행한 하위 분석에서 딥러닝 모델은 재관류 성공 여부에 대한 정보 없이도 최종 뇌경색 병변을 예측하는 데 있어 상업용 소프트웨어와 대등하거나 더 우월한 성능을 보여주었다.
5) 합병증 및 예후 예측(prediction of final lesion and complications)
뇌경색 환자에서의 출혈 변환은 정도에 따라 환자의 임상 증상을 악화시킬 수 있는 비교적 흔한 합병증으로, 다양한 영상학적 특징이 출혈 변환을 예측하는 데 사용될 수 있다[95]. 최근에는 메타분석을 통해 다양한 뇌관류 CT 매개변수를 사용하여 출혈 변환을 중등도의 성능으로 예측할 수 있음이 확인되었다[96]. Yu 등[97]의 연구에서는 다양한 기계 학습 모델을 사용하여 deconvolution을 적용하지 않은 뇌관류 MRI 소스 영상을 직접 분석하여 출혈 변환을 정확도 0.837로 예측할 수 있었고, 딥러닝 기반으로 한 다중 기법 MRI와 임상변수를 결합하여 만든 모델은 영상만을 이용한 모델에 비해 더 향상된 정확도를 보여 기계 학습이 출혈 변환 예측에 유용하게 활용될 수 있음을 입증하였다[98,99].
급성 뇌경색 환자의 예후를 radiomics를 이용하여 예측하고자 하는 시도들이 이루어지고 있다. 다양한 연구 결과에 따르면, MRI의 radiomics를 기반으로 한 기계 학습 모델은 mRS 3점 이상의 불량한 기능적 예후를 효과적으로 예측할 수 있었고, radiomics와 임상 정보를 결합한 모델은 radiomics 혹은 임상 정보 단독에 의한 모델보다 더 나은 예측력을 보였다[100].
3. 뇌경색 환자에서 임상 정보를 기반으로 한 기계 학습 모델 활용
뇌졸중 환자의 치료 결정 및 예후 예측에는 환자의 연령, 성별, 동반 질환, 환자의 병전 기능적 상태, 초기 뇌졸중 중증도 등 다양한 임상 변수들이 사용된다. 그러나 이러한 다양한 임상 변수를 종합적으로 고려하여 예측 모델을 만들 때 연구자가 기존에 인과관계가 확인되거나 의심되는 변수들을 선정하고 결과를 수기로 정리해야 하는 과정이 필요하기 때문에 분석 대상자 수가 많거나 변수가 많은 경우 예측 모델을 개발하는 것이 어려울 수 있다. 따라서 최근에는 뇌경색 환자에서 자연어 처리 과정(natural language processing, NLP)을 사용하여 비구조화된 문자 기반의 전자의 무기록이나 영상 결과 보고서를 직접 기계 학습에 활용하여 뇌경색 진단, 분류 및 임상적 예후를 예측하는 연구들이 진행되고 있다[101,102]. NLP는 비구조화된 의료 정보를 구조화된 데이터로 변환하고, 기계 학습 알고리즘을 거쳐 뇌졸중 환자의 치료 및 예후에 대한 의사 결정을 지원하는 데 활용될 수 있다.
1) 뇌경색의 진단과 병인 분류
뇌졸중의 진단에는 영상 정보와 함께 환자의 증상과 검사 정보를 포함한 임상 정보도 중요하기 때문에 이를 함께 활용하여 뇌졸중과 유사한 질환을 감별하고 뇌경색 및 일과성 허혈발작(transient ischemic attack, TIA)을 진단 및 분류하고자 하는 시도가 있었다[12,103,104]. 특히 조직학적 정의에 의한 일과성 허혈발작은 MRI 영상에서 병변이 확인되지 않을 수 있으며, 주로 환자의 임상 정보에 의해 진단을 하게 되므로 진단 보조 수단에 대한 필요성이 더 높다. Stanciu 등[104]은 전자의무기록에서 추출한 환자의 임상 증상, 인구학적 정보, 동반 질환 등의 정보를 기반으로 recursive feature elimination (RFE)과 least absolute shrinkage and selection operator (LASSO) 방법을 이용하여 일과성 허혈발작을 진단하는 모델을 개발하였고, 해당 모델은 기존의 ABCD2 점수 및 diagnosis of TIA (DOT) 점수에 비해 비슷하거나 더 나은 예측력을 보였다. 이처럼 초기 환자 진료 기록을 바탕으로 기계 학습 모델을 활용한 의사결정 보조 시스템이 만들어지면 응급실에서 진단과 치료 지연을 줄이는 데 도움이 될 수 있을 것이다[105].
뇌경색은 다양한 원인에 의해 발생하며, 뇌경색의 병인을 정확하게 분류하는 것은 이차 예방 전략을 세우는 데 있어서 중요하다. 최근에는 기계 학습을 이용하여 뇌경색의 원인을 감별하고자 하는 여러 연구들이 진행되고 있으며, Garg 등[101]은 뇌경색 환자의 전자 의무기록에서 특성(features)들을 추출하여 trial of Org10172 in acute stroke (TOAST) 분류가 가능함을 보여주었고, Guan 등[102]은 진단 정보, 시술 코드, 심초음파 결과 보고서 등을 기반으로 NLP를 활용하여 심장색전성 뇌경색을 92.2%의 정확도로 예측하였다. 최근에는 원인 불명의 뇌경색 환자에서 숨겨진 심장색전성 뇌경색을 감별하고자 하는 연구도 진행되고 있으며[106,107] 이러한 연구들을 통해 다양한 기계 학습 모델은 의료 자원이 부족한 지역에서 뇌졸중 환자를 정확하게 진단하고 예후를 예측하는 데 기여할 수 있을 것이다.
2) 뇌경색 후 임상적 예후 예측
뇌졸중 환자는 다양한 경과를 보일 수 있기 때문에 위험도 평가는 중요하며, 이를 통해 뇌졸중 환자의 치료 방향을 결정하고 의료 자원을 최적으로 활용할 수 있다. 현재 임상에서는 급성 뇌경색 이후의 예후를 예측하기 위해서 acute stroke registry and analysis of Lausanne (ASTRAL) score, iSCORE 등 다양한 점수 체계가 사용되고 있다[108,109]. 이러한 점수 체계는 대개 전통적인 통계 분석 모델을 통해 개발되었고, 뇌경색 발생 후 예후를 예측하는 데 비교적 높은 예측력을 보이고 있다.
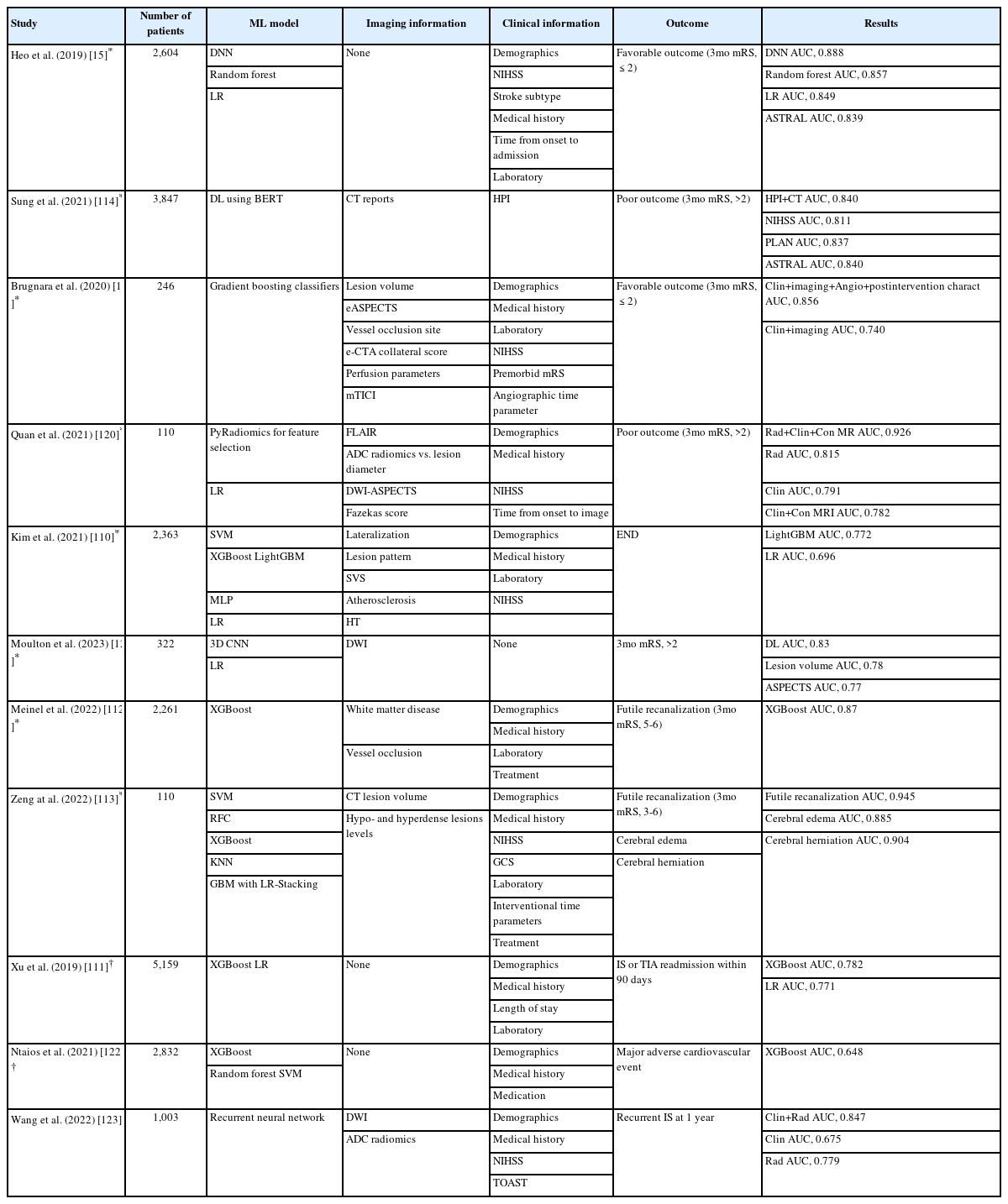
뇌경색 환자의 임상적 예후에는 입원 중 신경학적 증상 악화, 퇴원 이후 기능적 상태, 사망 및 재발이 포함되며 고전적인 점수 체계보다 더 높은 예측 정확성을 얻기 위해 기계 학습이 활용될 수 있다. 실제로 뇌경색 환자에서 입원 중 신경학적 증상의 악화, 뇌졸중 발생 후 3개월 시점의 mRS로 측정되는 퇴원 후 기능적 상태, 재입원이나 재발, 일상생활 정도 등을 예측하는 다양한 기계 학습 모델이 개발되고 있다[15,110,111]. 뿐만 아니라 다양한 임상 변수를 포함하여 학습시킨 기계 학습 모델은 기계적 혈전제거술 후 3개월째 불량한 예후, 악성 뇌부종, 뇌탈출 등을 예측할 수 있었으며 연령, 뇌경색 중증도, 증상 발생 후 입원까지 및 천자 후 재개통까지의 시간 등의 인자가 예후에 영향을 미치는 것을 보고하였다(Table 3) [112,113].
최근의 연구들은 특정 기계 학습 모델 및 딥러닝 모델이 기존의 통계 모델보다 뇌경색 후 환자의 임상적 예후를 더 정확하게 예측할 수 있음을 입증하고 있으며[15,114], 이와 함께 예후 예측에 영향을 줄 수 있지만 기존에 알려지지 않았던 인자들을 확인하여 뇌졸중에 대한 우리의 이해를 향상시키고 있다.
4. 뇌졸중 환자에서 기계 학습 모델 적용의 한계
의료 현장에서 기계 학습 활용의 중요성은 점점 더 증가하고 있으며, 다양한 임상 정보와 영상 정보를 기반으로 한 기계 학습 모델의 활용으로 향후에는 뇌졸중 환자 개개인의 특성을 고려한 맞춤형 치료가 가능해질 것으로 기대된다. 그러나 기계 학습 모델이 뇌졸중 진료 현장에서 널리 사용되기 전에 해결해야 할 몇 가지 과제들이 있다. 첫째, 기계 학습 모델의 성능은 데이터의 양과 질에 크게 의존한다. 현재 뇌졸중 환자에서 기계 학습을 활용한 연구들은 대부분 소규모의 데이터셋을 사용하여 기계 학습 모델이 기존의 진단 방법에 비해 열등하지 않거나 조금 더 나은 정확도를 보여주는 데 중점을 두고 있다. 이는 데이터의 전처리 과정이 복잡하고, 환자의 영상 정보와 임상 정보의 형식이 다양하여 표준화가 어렵기 때문에, 이를 통해 생성된 모델을 다른 데이터셋에 적용하는 데 한계가 있기 때문이다. 둘째, 뇌졸중은 발병 후 시간 및 치료 여부에 따라 증상과 예후가 변화할 수 있는 질환이기 때문에 이러한 시간의존적 효과를 모델에 고려하는 것이 중요하다. 마지막으로 기계 학습을 이용한 많은 뇌졸중 연구가 기존에 알려진 특성들을 추출하여 분류 혹은 예측 모델을 개발하는 방식으로 이루어지고 있다. 이미 많은 연구에서 기계 학습을 이용한 예측 모델이 전문가의 예측과 대등하거나 오히려 더 나은 예측을 보여주는 것이 확인되었으므로, 추후의 연구는 인간의 능력으로 예측이 어려웠던 복잡한 현상의 예측 및 기존에 알려지지 않은 결정 인자들을 찾기 위한 방향으로 진행되어야 할 것이다.
또한 기계 학습 자체가 가지는 문제점도 있다. 딥러닝을 포함한 최근의 기계 학습 모델은 복잡한 인공 신경망을 사용함으로써 결과를 도출하지만 결과를 생성하는 과정을 제시하고 설명할 수 없다는 한계가 있다. 특히 의료 영역에서는 이러한 블랙박스 문제가 기계 학습의 결과를 실제 진료 현장에 적용하는 데 장애가 될 수 있다. 최근에는 기계 학습 모델이 가지고 있는 이러한 단점을 보완하기 위해 설명 가능한 인공지능(explainable artificial intelligence)을 활용한 연구가 이루어지고 있으며, class activation map (CAM), gradient-weighted CAM (Grad-CAM), trainable attention 등 다양한 방법을 이용하여 결과에 이르는 결정 단계를 유추하고 설명하려는 시도가 이루어지고 있다[115,116].
뇌졸중 분야에서 생성형 인공지능(generative artificial intelligence)을 활용한 연구는 아직 많이 이루어지지 않고 있다. 생성형 인공지능은 이미지, 음성, 텍스트 등의 데이터 생성과 수정에 매우 효과적으로 활용될 수 있으며, 특히 의료 영상 데이터에 대한 유용성이 크게 기대되고 있다[117]. 예를 들어, 생성형 인공지능 모델을 통해 뇌졸중의 다양한 단계와 유형에 대한 합성 CT나 MRI 이미지를 생성할 수 있다. 이런 합성 데이터는 진단과 치료제 개발에 도움이 될 뿐만 아니라, 관련 연구를 위한 학습 데이터로도 사용될 수 있다. 또한, 생성형 인공지능 모델은 뇌졸중 환자의 다양한 치료 결과를 시뮬레이션 하는 데 사용될 수 있어, 환자마다 가장 적합한 치료 전략을 결정하는 데 도움이 될 수 있다. 하지만 생성형 인공지능 모델은 기존 기계 학습 방법에 비해 훨씬 많은 양의 데이터와 복잡한 모델링을 필요로 하며, 모델이 어떻게 작동하는지 해석하기 어려울 수 있다.
따라서 향후 뇌졸중 환자 진료 분야에서 기계 학습을 활용하기 위해서는 양질의 많은 데이터를 수집하고 표준화하여 모델을 훈련시키는 것이 필수적이다. 또한 기존 연구 방법의 한계를 극복할 수 있도록 설명 가능한 인공지능뿐만 아니라 생성형 인공지능을 포함한 다양한 기계 학습 방법론에 대한 연구가 필요하다.
결 론
뇌경색 환자 진료에 있어서 기계 학습 및 인공지능 기술의 활용은 아직 초기 단계에 머물러 있지만, 현재 많은 연구들이 진행되고 있으며 매우 흥미로운 발전을 보여주고 있다. 이러한 기술의 활용은 향후 뇌졸중 진료에 있어서 정확하고 빠른 진단, 환자에게 맞춤화된 치료 계획 수립, 환자의 예후 예측 및 개선에 많은 도움을 주며 환자의 건강과 질병 관리에 긍정적인 영향을 줄 것으로 기대된다.
Acknowledgements
This research was supported by Ansan-Si hidden champion fostering and supporting project funded by Ansan city.